Replicación (soluciones)
1.- ¿En qué consiste la replicación del ADN? ¿Cuáles son sus características más importantes?
• La replicación o duplicación del ADN es el proceso que permite, a partir de una molécula de ADN (doble hélice), sintetizar dos idénticas. Es decir: ADN —› 2 ADN.
• La replicación es semiconservativa (semiconservadora), o sea, que cada doble hélice resultante está formada por una de las cadenas iniciales y otra de nueva formación.
• Es un proceso bidireccional, pues cuando las dos cadenas iniciales empiezan a separarse, formando la llamada burbuja de replicación, las horquillas de ambos lados avanzan en sentidos opuestos respecto a sus orígenes.
• Es unifocal en procariotas y multifocal en eucariotas, lo cual quiere decir que existe un único origen de replicación en el ADN circular de las células procariotas, mientras que en cada uno de los cromosomas lineales de las eucariotas hay múltiples orígenes.
2.- ¿A qué se llama origen de replicación? ¿Qué es oriC?
La replicación no comienza al azar ni en cualquier parte. Un complejo sistema de regulación intracelular controla el momento del ciclo celular para iniciar el proceso, siendo preceptivo el reconocimiento enzimático de una determinada secuencia de nucleótidos, llamada origen de replicación.
El origen de replicación es el lugar del cromosoma donde, como su propio nombre indica, se inicia la replicación del ADN.
Los cromosomas bacterianos (y los plásmidos) suelen tener un solo origen de replicación, mientras que los eucariotas poseen numerosos orígenes por cromosoma (a veces, miles), posibilitando que el genoma se pueda replicar en muy poco tiempo.
El origen de replicación de la bacteria Escherichia coli está constituido por una secuencia de 245 pares de bases y se conoce como oriC.
3.- ¿Cuáles son las enzimas principales que participan en la replicación del ADN?
• Las helicasas, que son las encargadas de separar las dos cadenas del ADN que se va a replicar, deshaciendo los puentes de hidrógeno entre las bases complementarias, actividad que requiere el aporte energético del ATP.
• Las topoisomerasas solucionan los problemas del tensionado de la doble hélice (derivado de la actividad de las helicasas).
• Las primasas son ARN polimerasas encargadas de sintetizar un oligonucleótido, habitualmente ARN, llamado iniciador, cebador o primer, proporcionando un extremo 3’-OH necesario para la actividad de las ADN polimerasas.
• Las ADN polimerasas tienen como función principal la elongación de las nuevas cadenas de ADN, incorporando un nucleótido cada vez al extremo 3’-OH.
• Las ADN ligasas se encargan de la unión de los fragmentos de ADN adyacentes mediante enlace fosfodiéster.
Nota.- Algunos autores denominan replisoma al conjunto enzimático fundamental para realizar el proceso de replicación del ADN, que incluye helicasas, topoisimerasas, primasas, polimerasas y ligasas.
4.- ¿Qué papel desempeñan en la replicación las llamadas proteínas SSB(single stranded binding protein)?
Cuando las helicasas separan la doble hélice del ADN, unas proteínas no enzimáticas, conocidas como SSB, se unen a las cadenas sencillas impidiendo el restablecimiento de los puentes de hidrógeno mientras se completa el proceso de replicación.
5.- ¿Puede concretar lo que precisa la ADN polimerasa para desempeñar su función?
• Los 4 desoxirribonucleósidos trifosfato (dNTP), o sea: dATP, dGTP, dCTP, dTTP.
• Una cadena de ADN que sirva como molde para copiar, según la complementariedad de bases.
• Un oligonucleótido (por lo común, ARN), llamado cebador, que proporcione el extremo 3’-OH, necesario para que la ADN polimerasa pueda iniciar la síntesis de ADN.
• En tales circunstancias, la ADN polimerasa actúa continuamente adicionando un nucleótido al extremo 3’-OH, de forma que la nueva cadena va creciendo en la dirección 5’—› 3’.
6.- Si la ADN polimerasa sintetiza en la dirección 5’—› 3’, ¿qué problema plantea el antiparalelismo de las cadenas del ADN?
La cuestión fue aclarada por R. Okazaki, quien observó trozos pequeños de ADN cuando se ultimaba la síntesis.
Este descubrimiento permitió establecer que una de las cadenas, llamada conductora, se sintetizaba de manera continua y en la misma dirección que avanza la horquilla de replicación. La síntesis de la otra cadena tenía lugar en dirección contraria y de modo discontinuo, originando trozos de unos mil nucleótidos, llamados fragmentos de Okazaki, la unión de los cuales, mediante la acción de la ADN ligasa, dará lugar a la cadena retrasada.
7.- Busque en Internet: “EC 2.7.7.7”. Indique su nombre, la clase y la función que realiza en tres ocasiones sucesivas.
Se trata de la enzima ADN polimerasa dependiente de ADN. Pertenece a la clase 2 (Transferasas).
Utiliza como sustrato los 4 desoxinucleósidos trifosfato (dNTP): dATP, dGTP, dCTP, dTTP. Su función es separar dos fosfatos del sustrato (ruptura que además proporciona el aporte energético) y transferir el nucleósido monofosfato al extremo 3’-OH, estableciendo el enlace fosfodiéster:
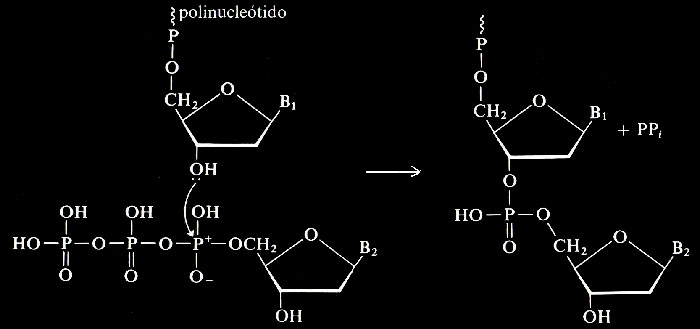
(Al estar representado sólo el polinucleótido que se sintetiza, pero no la cadena molde de ADN, no se refleja que las bases B1 y B2 quedan emparejadas con sus correspondientes en el ADN molde, según la complementariedad: A con T y G con C).
La ADN polimerasa cataliza sucesivamente la siguiente reacción:
dNTP + ADNn —› ADNn+1 + PPi (difosfato)
dNTP + ADNn+1 —› ADNn+2 + PPi
dNTP + ADNn+2 —› ADNn+3 +PPi
El difosfato o pirofosfato (PPi) es hidrolizado por una pirofosfatasa, de forma que el equilibrio se desplaza hacia la derecha.
8.- ¿Puede aclarar esta frase: “Las ADN polimerasas requieren un cebador”?
Las ADN polimerasas no pueden iniciar la síntesis de novo, es decir, no pueden actuar si no hay un grupo 3’-OH para formar el enlace fosfoéster con el fosfato de la posición α, quedando éste separado de los otros dos.
Cada dNTP está formado por 3 grupos fosfato (α, β, γ), desoxirribosa (dR) y la base (B). O sea, de modo abreviado: P(γ)-P(β)-P(α)-dR-B.
…3’C- O H + H O - P(α)…(dNTP) —› H2O + …3’C-O-P(α)…(dNMP) + PPi
Sin el grupo 3’-OH no se podría establecer dicho enlace, aunque también es imprescindible una cadena molde de ADN.
9.- Busque en Internet (o en su libro) para ilustrar con fórmulas, estando presente la cadena de ADN molde, la reacción que cataliza la ADN polimerasa y añada un breve comentario.
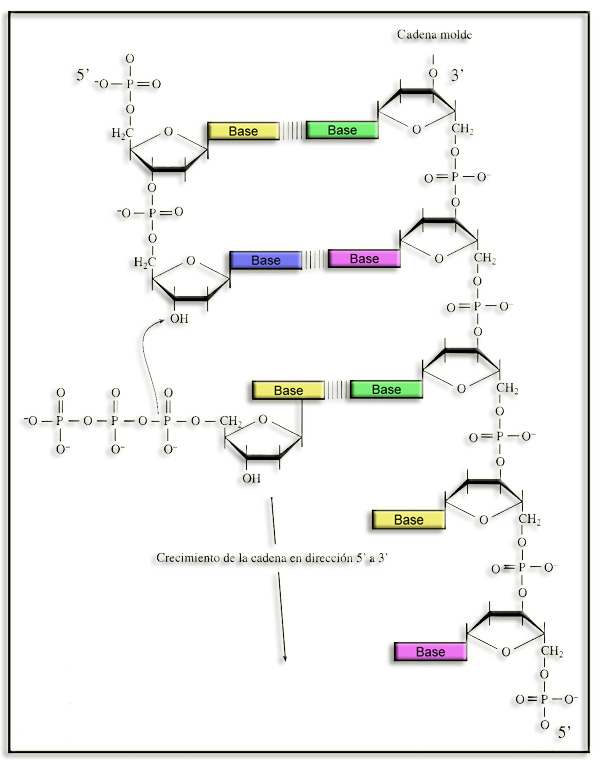
Biología 2 McGraw-Hill (2001)
Se observa que la importancia del extremo 3’-OH es determinante puesto que posibilita la formación del nuevo enlace. De esta forma queda separado el difosfato y establecido el enlace fosfodiéster con el nucleótido incorporado, cuya base debe ser complementaria (A con T y G con C) con la de la cadena molde. La nueva cadena se sintetiza en dirección 5’—› 3’.
10.- ¿Permanece el ARN iniciador (cebador) en las cadenas de ADN que se han sintetizado?
El oligonucleótido iniciador, habitualmente ARN, es eliminado posteriormente por acción exonucleasa de la ADN polimerasa I. El hueco correspondiente se rellena mediante la actividad sintetizadora de la misma. Esta enzima posee centros activos diferentes para tales actividades.
11.- ¿Podría comenzar la actividad de la ADN polimerasa sin el extremo 3’-OH del oligonucleótido iniciador?
Existen ADN lineales que contienen una proteína terminal unida covalentemente, la cual interviene como iniciadora al proporcionar el OH de un aminoácido (serina, treonina o tirosina). En estos casos la ADN polimerasa reconoce también el OH del aminoácido y lo utiliza para establecer previamente un enlace fosfoéster con el primer desoxirribonucleótido y proseguir la síntesis de ADN.
Este excepcional mecanismo de iniciación mediante proteína terminal tiene lugar en la replicación de algunos virus bacterianos y adenovirus.
12.- ¿Qué quiere decir que la ADN polimerasa tiene una fidelidad situada en torno a 10-8?
Significa que la ADN polimerasa introduce un nucleótido equivocado por cada cien millones (108) que se han incorporado.
Cabe señalar que la replicación del ADN es un proceso extraordinariamente fiel, posibilitando que la información genética se transmita de una generación a otra sin errores o con el menor número de ellos.
Nota.- Hay textos que indican una fidelidad de 10-9, e incluso, 10-10
13.- ¿Cómo puede ser tan alta la fidelidad de la ADN polimerasa si posee el mismo centro activo para la inserción de cualquiera de los 4 desoxirribonucleótidos?
Es fundamental la presencia de la cadena molde de ADN para dirigir y controlar todo el proceso. La enzima ADN polimerasa inserta el nuevo nucleótido cuando la base de éste queda emparejada correctamente con la correspondiente del ADN molde (A con T y G con C).
En otras palabras: la posibilidad de incorporación del nucleótido que ocupa el centro activo es casi nula si no puede emparejarse con el de la hebra molde.
14.- Cite algunos mecanismos celulares para corregir los posibles errores en la replicación del ADN.
La célula posee diversos mecanismos tendentes a solucionar estos errores: reparación de apareamientos incorrectos, reparación por eliminación de bases o de nucleótidos, reparación directa del ADN.
15.- Exponga una interpretación del siguiente esquema:
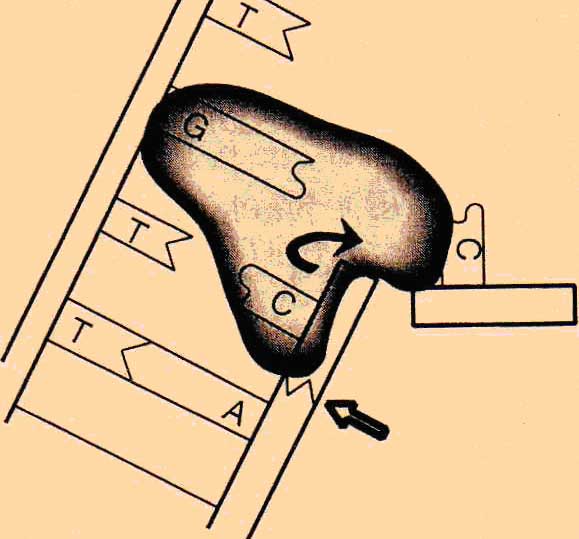
Este esquema representa la corrección de errores o lectura de prueba efectuada por la propia ADN polimerasa. Se observa un emparejamiento incorrecto, TC. La flecha recta marca la rotura de la cadena, para quitar C y poner A, todo ello antes de insertar el nucleótido del siguiente par, GC.
16.- ¿Qué son las unidades de replicación o replicones?
En cada cromosoma de las células eucariotas hay muchos orígenes de replicación. A ambos lados de los mismos se van formando las correspondientes burbujas de replicación, que se van ampliando progresivamente de manera bidireccional, constituyendo las unidades de replicación o replicones.
Evidentemente, todas las burbujas de replicación acabarán por unirse al finalizar la réplica de todo el ADN del cromosoma.
17.- ¿Por qué se dice que la horquilla de replicación es asimétrica?
La llamada burbuja de replicación empieza a abrirse en el origen de replicación. En cada extremo de dicha burbuja se forma una horquilla de replicación, de manera que las dos hebras se van copiando simultáneamente.
Todas las ADN polimerasas sintetizan en dirección 5’—› 3’. Por consiguiente, una de las dos hebras, la llamada cadena continua o conductora, es de crecimiento continuo puesto que la horquilla se va abriendo en el mismo sentido que la ADN polimerasa añade los nucleótidos. Mientras que la otra hebra, llamada cadena discontinua o retrasada, debe sintetizarse por partes y en sentido opuesto, alejándose de la horquilla. Tales partes, de unos 1000 nucleótidos, reciben la denominación de fragmentos de Okazaki, los cuales van siendo unidos mediante la acción de la ADN ligasa.
18.- Busque en Internet: “EC 6.5.1.1”. Indique su nombre, la clase y la reacción que cataliza.
ADN ligasa. Pertenece a la clase 6 (Ligasas). Su función es unir dos fragmentos de ADN utilizando como fuente energética ATP, que se hidroliza en AMP y PPi (difosfato):
ATP + ADNn + ADNm —› AMP + PPi + ADNn+m
19.- Preste atención a este texto: “La replicación del ADN está catalizada por una enzima que se denomina ADN polimerasa. Las ADN polimerasas sólo funcionan utilizando una cadena de ADN como molde, necesitando inexcusablemente un cebador”. ¿Por qué se emplea en muchos libros el singular y el plural, indistintamente, al referirse a dicha enzima?
Se trata de una simplificación expresiva, pues en realidad hay diferentes formas de ADN polimerasa.
En la bacteria E. coli se han descrito tres ADN polimerasas (I, II, III). La III y la I se encargan de la replicación y de la corrección de errores. La II realiza la reparación del ADN dañado por determinados agentes físicos.
La ADN polimerasa III es la que sintetiza la mayor parte del nuevo ADN y a ella suele ir dirigido el modo expresivo en singular.
Mediante modernos análisis estructurales se ha observado que las ADN polimerasas tienen una geometría espacial bastante similar, en forma de mano derecha (semicerrada), con tres dominios claramente diferenciados (dedos, palma y pulgar).
Nota.- La llamada ADN polimerasa I fue aislada por primera vez por Arthur Kornberg en 1955, quien compartió en 1959 el Premio Nobel de Medicina con Severo Ochoa, que había logrado la primera síntesis de ARN in vitro.
20.- ¿Cuáles son las ADN polimerasas de los eucariotas?
En eucariotas hay distintas formas de polimerasas: α, β, γ, δ, ε. Estas 5 variantes de ADN polimerasa se reparten todas las tareas de la elongación y corrección de errores.
La ADN polimerasa alfa (α) actúa también como primasa y parece intervenir en la síntesis de la cadena retrasada. La γ interviene en la replicación del ADN mitocondrial. La ADN polimerasa δ podría sintetizar la cadena conductora. Algunos autores apuntan que la llamada ADN polimerasa ε participa en la reparación del ADN.
Nota.- En 2º de Bachillerato se suele recomendar la denominación genérica de ADN polimerasa y obviar las sinonimias, que, dicho sea de paso, tienen asignada por la Comisión de Enzimas la misma numeración (EC 2.7.7.7):
Other name(s): DNA polymerase I; DNA polymerase II; DNA polymerase III; DNA polymerase α; DNA polymerase β; DNA polymerase γ; DNA nucleotidyltransferase (DNA-directed); (…)
21.- ¿Qué representa el esquema adjunto? Nombre las partes numeradas e indique la polaridad en los extremos marcados (*).
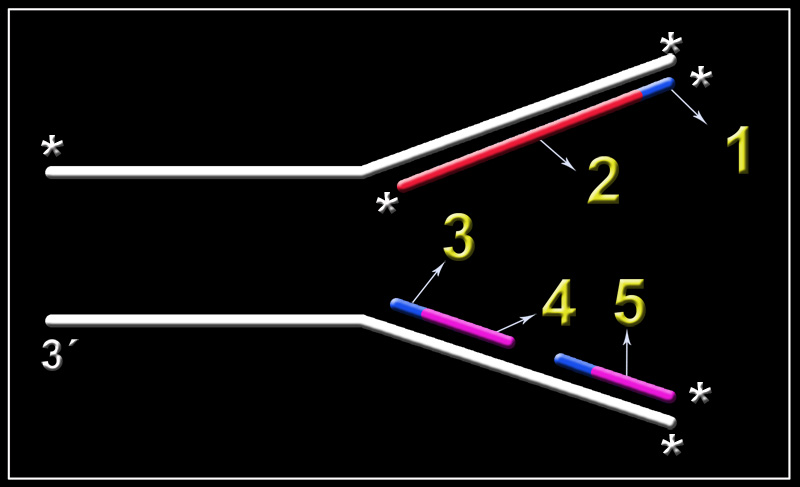
El esquema corresponde a una horquilla de replicación.
1 = oligonucleótido iniciador (cebador). 2 = cadena continua o conductora. 3 = cebador. 4 = 2º fragmento de Okazaki. 5 = 1er fragmento de Okazaki.
La polaridad de los lugares marcados es la siguiente:
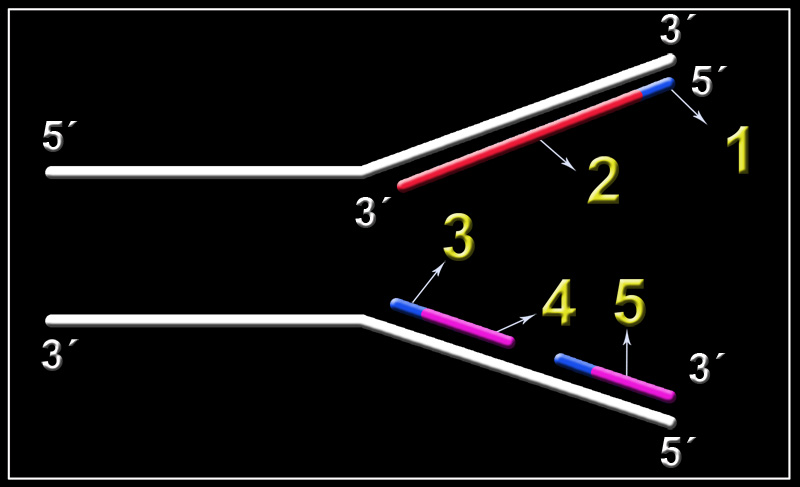
22.- Complete el esquema adjunto, nombre las partes numeradas e indique la polaridad en los lugares marcados (*).
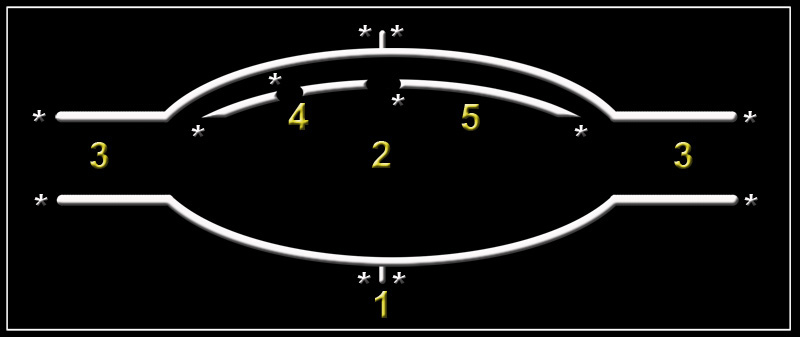
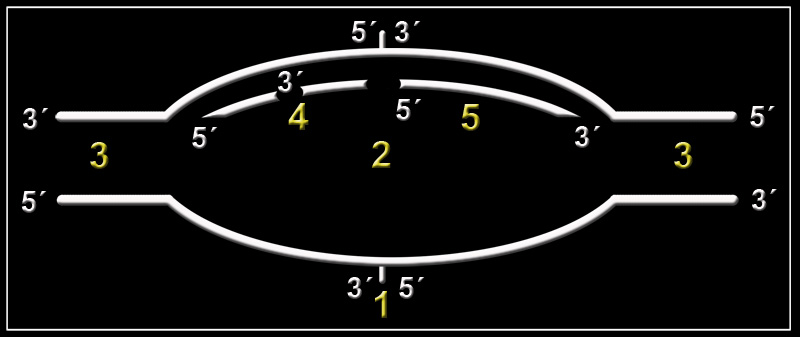
1 = origen de replicación. 2 = burbuja de replicación. 3 = ADN molde. 4 = fragmentos de Okazaki (cadena retrasada). 5 = cadena continua o conductora (la dirección de síntesis es 5’—› 3’).
Dibujo completo y polaridad:
23.- Nombrar las partes numeradas de la horquilla de replicación adjunta. ¿Qué función desempeña lo indicado con el número 13?
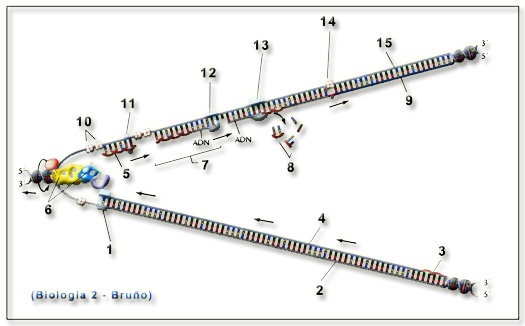
He aquí el esquema completo:
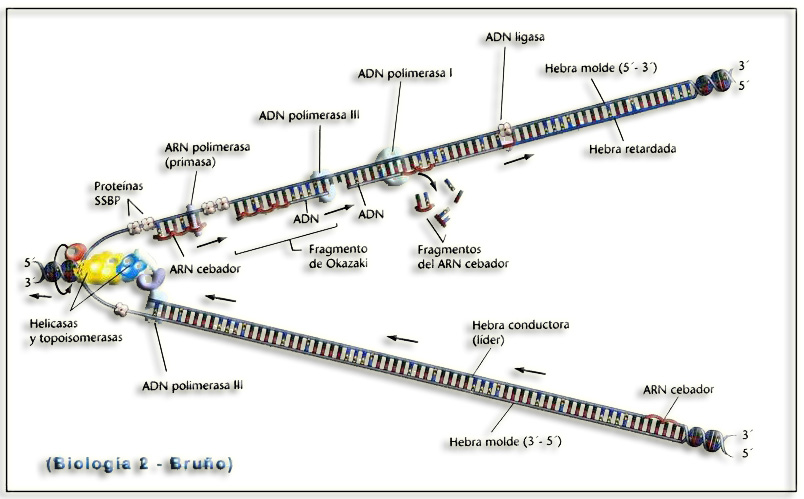
La ADN polimerasa I (marcada en la pregunta con el nº 13) presenta actividad exonucleasa y elimina el ARN cebador (primer) de cada fragmento. El hueco que queda es rellenado por la propia ADN polimerasa I, que sintetiza ADN (5’—› 3’) con una secuencia complementaria a la del ADN molde.




