Traducción 2 (soluciones)
1.- ¿Cuál es la señal del ARN mensajero que marca el inicio de la traducción?
La señal de inicio utilizada en la traducción es un triplete AUG (5’-AUG-3’), denominado codón iniciador, que se halla muy próximo al extremo 5’ del ARN mensajero.
Nota.- De forma excepcional, los procariotas pueden emplear como señales de inicio los tripletes GUG o UUG.
2.- ¿Cuál es el aminoácido que figura al comienzo de la cadena polipeptídica sintetizada?
El codón iniciador AUG determina que todas las proteínas sintetizadas empiecen por el aminoácido metionina (M, Met) en las células eucariotas, o bien, por formilmetionina (fMet) en las procariotas (ver nota). Con frecuencia, el aminoácido inicial desaparece durante el procesado postraduccional.
Nota.- Cuando las células procariotas, excepcionalmente, utilizan como codón iniciador GUG o UUG (en vez de AUG), tiene lugar la incorporación de fMet en ambos casos.
La síntesis proteica en orgánulos eucarióticos tales como mitocondrias y cloroplastos también empieza con fMet.
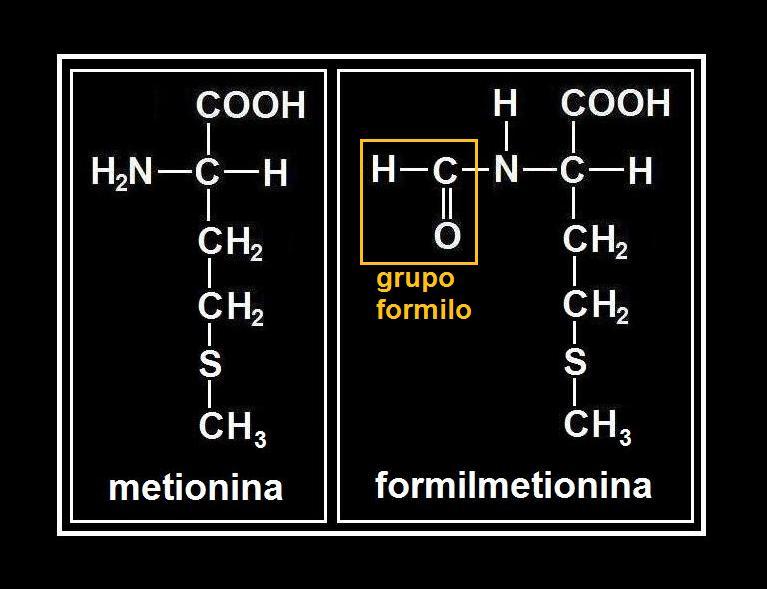
3.- Escriba la fórmula de los aminoácidos metionina y formilmetionina (consulte su libro).
Ambos son aminoácidos azufrados, como puede observarse en las fórmulas. La formilmetionina se caracteriza por la presencia de un grupo formilo (H-C=O) ligado al N de la metionina.
4.- Cite las fases o etapas que suelen considerarse en la traducción del ARN mensajero.
Las fases características o fundamentales de la traducción son tres: iniciación, elongación y terminación, cada una de las cuales requiere un aporte energético (ATP, GTP) y una serie factores proteicos específicos.
Cabe señalar que hay libros en los que el mecanismo de la traducción es desarrollado en cinco fases: activación de los aminoácidos, iniciación, elongación, terminación y maduración o procesado de la proteína.
Los autores que consideran 3 fases interpretan que otros procesos, tales como la activación y la maduración, tienen carácter complementario y no son estrictamente fases de la traducción.
5.- Escriba un resumen de la traducción considerando las tres etapas fundamentales (excluya la activación y la maduración).
El proceso de la traducción, omitiendo la activación de los aminoácidos y el procesado postraduccional (maduración), se puede resumir así:
• Iniciación. El comienzo de la traducción se caracteriza por la formación del complejo de iniciación (80 S en eucariotas, 70 S en procariotas), que está integrado por un ribosoma unido al ARNm y al ARNt iniciador cargado con el primer aminoácido (metionina en eucariotas, formilmetionina en procariotas).
Esta fase requiere aporte energético (GTP, ATP) y proteínas citosólicas llamadas factores de iniciación.
• Elongación. La cadena peptídica se va alargando por unión covalente de sucesivos aminoácidos (enlaces peptídicos), cada uno de los cuales es situado previamente en el ribosoma por su correspondiente ARNt, en función de los enlaces específicos que deben establecerse entre el codón (ARNm) y el anticodón (ARNt).
Cada ribosoma se va desplazando a lo largo del ARNm de tres en tres tripletes (translocación), en sentido 5’ --> 3’.
Esta fase precisa proteínas citosólicas llamadas factores de elongación y aporte energético (GTP).
• Terminación. El final de la síntesis proteica está señalado en el ARNm por uno de estos tres codones de terminación: UAA, UAG o UGA, llamados mudos o sin sentido. Tras la fijación de un factor de liberación a uno de ellos tiene lugar la separación de la cadena peptídica y de las subunidades ribosómicas, las cuales pueden reiniciar el proceso.
Esta fase requiere el concurso de factores de liberación.
6.- ¿Cuándo se dice que un ARN transferente está “cargado”?
Un ARNt se considera “cargado” cuando se ha unido a su aminoácido específico, estableciéndose un enlace tipo éster entre el 3’-OH del ARNt (extremo aceptor) y el grupo carboxilo del aminoácido, el cual queda “activado”.
El conjunto formado por la molécula de aminoácido unida a otra de ARN transferente recibe la denominación de aminoacil-ARNt.
Estrictamente, las uniones entre los aminoácidos y sus respectivos ARNt no son fases de la traducción, pero resultan absolutamente necesarias para posibilitar dicho proceso.
7.- ¿En qué consiste la activación de los aminoácidos? Escriba la reacción general (sin fórmulas).
• La activación de los aminoácidos proteicos consiste en la unión de cada uno de ellos a su ARN transferente específico, mediante acción enzimática, proceso que tiene lugar en el citosol (así como en el interior de cloroplastos y mitocondrias).
Estas reacciones de activación son catalizadas por un grupo de enzimas dependientes de ATP y Mg2+, denominadas genéricamente “aminoacil-ARNt ligasas”, de las cuales hay 20 distintas, una para cada aminoácido. Cada una de ellas es muy específica tanto para el aminoácido como para el ARNt respectivo (ver nota).
• La reacción general de activación es:
ATP + aminoácido + ARNt —› aminoacil-ARNt + AMP + PPi
El pirofosfato o difosfato (PPi) es hidrolizado a fosfato inorgánico, razón por la cual el proceso activador presenta un balance energético favorable.
De esta forma, cada aminoácido queda unido al extremo aceptor de un ARNt específico, resultando que un gran número de transferentes se encuentran enlazados covalentemente a sus aminoácidos respectivos antes de iniciarse la traducción.
Nota.- Las aminoacil-ARNt ligasas de las células eucarióticas presentan una propiedad “autocorrectora”. Dicha propiedad permite que, si se inicia una activación errónea, el proceso se invierta quedando el aminoácido impropio libre de nuevo.
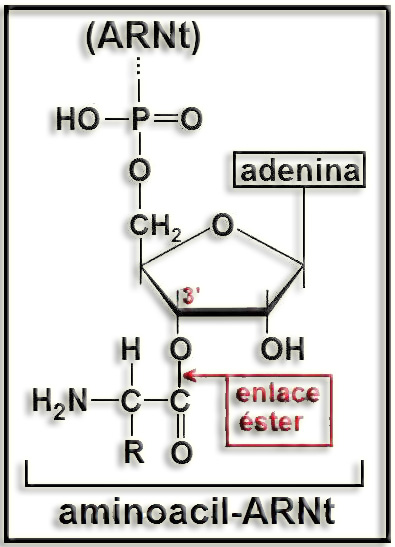
8.- ¿Qué tipo de enlace se establece entre el aminoácido y el ARN transferente? Haga un esquema.
• El aminoácido se une al ARNt mediante un enlace covalente tipo éster, quedando constituido el correspondiente aminoacil-ARNt. La unión se establece entre el grupo carboxilo del aminoácido y el llamado extremo aceptor del ARNt (que, generalmente, es la posición 3’-OH).
• Esquema:
9.- Escriba sin fórmulas la reacción de activación del aminoácido leucina (L, Leu) y el nombre de la enzima que cataliza el proceso.
• La reacción es:
ATP + leucina + ARNtLeu —› leucil-ARNtLeu + AMP + PPi
(O bien: ATP + leucina + ARNtL —› leucil-ARNtL + AMP + PPi)
• La enzima activadora es: leucina-ARNtLeu ligasa
10.- Escriba sin fórmulas la reacción de activación de los aminoácidos serina (Ser, S) y tirosina (Tyr, Y), así como el nombre de la enzima activadora correspondiente.
• Activación de la serina:
ATP + serina + ARNtSer —› seril-ARNtSer + AMP + PPi
Enzima activadora: serina-ARNtSer ligasa
• Activación de la tirosina:
ATP + tirosina + ARNtY —› tirosil-ARNtY + AMP + PPi
Enzima activadora: tirosina-ARNtY ligasa
11.- (Busque en Internet). Escriba solamente el nombre de las siguientes enzimas: “EC 6.1.1.1”, “EC 6.1.1.9” y “EC 6.1.1.11”.
• “EC 6.1.1.1” corresponde a la tirosina-ARNtY ligasa
• “EC 6.1.1.9” corresponde a la valina-ARNtV ligasa
• “EC 6.1.1.11” corresponde a la serina-ARNtS ligasa
Nota.- Las abreviaturas y letras mayúsculas correspondientes a estos aminoácidos son: Tyr, Y (tirosina); Val, V (valina); Ser, S (serina).
12.- (Internet: “EC 6.1.1.6”). Cite el nombre de la enzima que cataliza el proceso adjunto. Identifique los compuestos A, B, C, D, E y F. Escriba la reacción sin fórmulas.
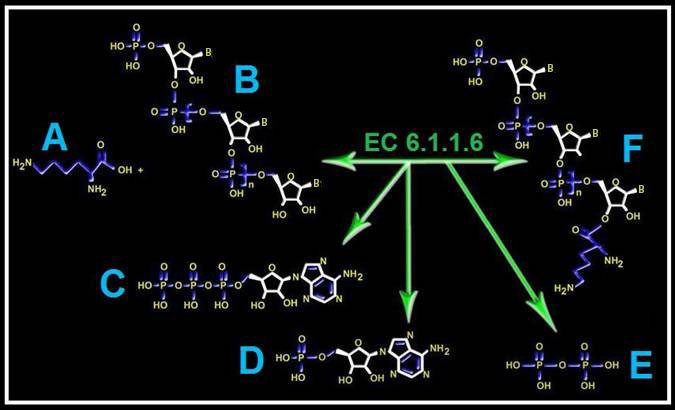
• “EC 6.1.1.6” corresponde a: lisina-ARNtLys ligasa
• Nombre de los compuestos:
A = lisina (Lys, K)
B = ARN transferente específico de la lisina: ARNtLys (ARNtK)
C = adenosín trifosfato (ATP)
D = adenosín monofosfato (AMP)
E = difosfato o pirofosfato (PPi)
F = lisil-ARNtLys (lisil-ARNtK)
• La reacción sin fórmulas es:
![]()
13.- (Internet: “EC 6.1.1.11”). Cite el nombre de la enzima que cataliza el proceso adjunto. Identifique los compuestos A, B, C, D, E y F. Escriba la reacción sin fórmulas.
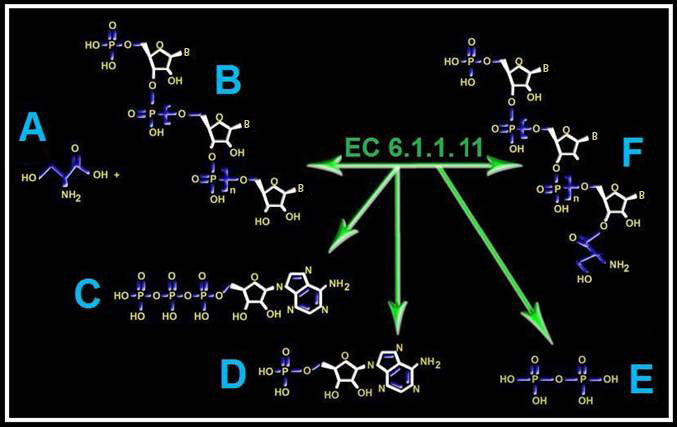
• “EC 6.1.1.11” corresponde a: serina-ARNtSer ligasa
• Nombre de los compuestos:
A = serina (Ser, S)
B = ARN transferente específico de la serina: ARNtSer (ARNtS)
C = adenosín trifosfato (ATP)
D = adenosín monofosfato (AMP)
E = difosfato o pirofosfato (PPi)
F = seril-ARNtSer (seril-ARNtS)
• La reacción sin fórmulas es:
![]()
14.- Escriba sin fórmulas la reacción de activación catalizada por “EC 6.1.1.9” (busque en Internet).
• “EC 6.1.1.9” corresponde a la enzima: valina-ARNtV ligasa
• La reacción de activación es la siguiente:
![]()
15.- ¿Por qué se dice que las enzimas aminoacil-ARNt ligasas son doblemente específicas? Escriba la reacción global.
• Cada aminoacil-ARNt ligasa resulta doblemente específica porque, por un lado, debe reconocer a uno de los 20 aminoácidos, y por otro, a un tipo concreto de ARN transferente (gracias a una decena de nucleótidos referenciales situados en posiciones críticas, que difieren de un ARNt a otro).
• La reacción global que catalizan dichas enzimas es la siguiente:

Este proceso requiere la presencia de iones Mg2+ y está favorecido por la hidrólisis del pirofosfato:
PPi + H2O —› 2 Pi + energía
16.- ¿Por qué se dice en algunos libros que la reacción de activación de los aminoácidos ocurre en dos etapas? (Aclare la respuesta con esquemas).
La acción enzimática de cada aminoacil-ARNt ligasa determina, en primer lugar, que el aminoácido se una al AMP, quedando enlazado seguidamente con su ARN transferente específico, formando el aminoacil-ARNt.
Esquemáticamente (E = enzima, a = aminoácido):
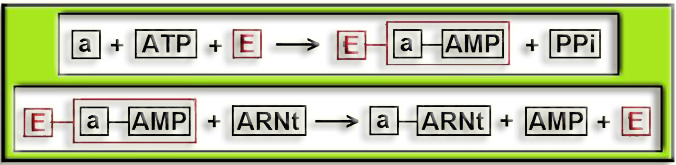
Se observa que en la 1ª etapa se forma un aminoacil-AMP unido a la enzima, siendo en la 2ª cuando tiene lugar la transferencia al ARNt para formar el aminoacil-ARNt.
17.- En relación con la pregunta anterior, haga un esquema de la estructura molecular de los “aminoacil-AMP” y “aminoacil-ARNt”.
• La estructura molecular de los aminoacil-AMP (adenilatos de aminoacilo), que se forman en el centro activo de las enzimas aminoacil-ARNt ligasas, es la siguiente:
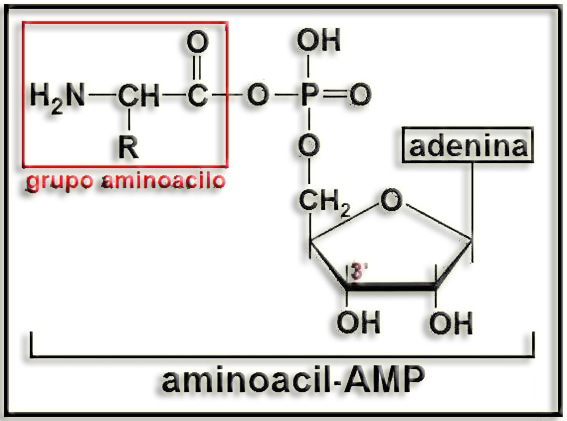
• La acción enzimática determina, seguidamente, que el grupo aminoacilo sea transferido al extremo aceptor del ARNt, quedando constituido el aminoacil-ARNt:
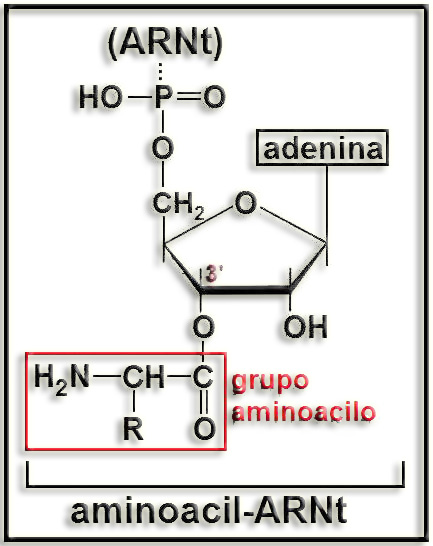
Se observa que el aminoácido queda unido al ARNt mediante un enlace covalente tipo éster.
18.- En relación con la unión de los aminoácidos a sus correspondientes ARN transferentes, ¿qué esquema le parece más correcto, A o B?
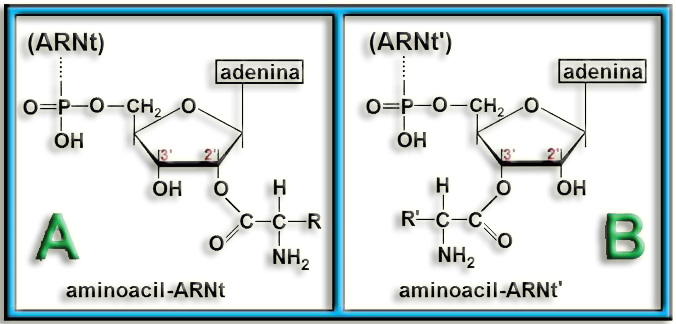
En principio puede considerarse que ambos esquemas son correctos, pues según la posición concreta de la esterificación se distinguen dos subtipos de enzimas aminoacil-ARNt ligasas:
• Subtipo I: la unión del aminoácido tiene lugar en el hidroxilo 2’ del nucleótido terminal del ARNt (figura A), pero seguidamente es trasladado a la posición 3’ por isomerización.
• Subtipo II: la esterificación se produce directamente sobre el hidroxilo 3’ del correspondiente ARNt (figura B).
Nota.- Las enzimas del subtipo I son monoméricas y reconocen los aminoácidos grandes y polares. Las del subtipo II son diméricas o tetraméricas y actúan con los aminoácidos más pequeños e hidrófobos.
19.- Internet: “EC 2.3.2.12”. ¿Cuál es su nombre y qué función realiza?
• “EC 2.3.2.12” corresponde a: peptidil transferasa (ver nota).
• Su función es catalizar la formación de los enlaces peptídicos durante la traducción de ARN mensajero. Gracias a esta acción enzimática, la cadena peptídica se va alargando por adición de sucesivos aminoácidos, según la reacción:
peptidil-ARNt(1) + aminoacil-ARNt(2) —> ARNt(1) + peptidil-aminoacil-ARNt(2)
Nota.- El complejo enzimático peptidil transferasa posee varios componentes proteicos, pero en la catálisis intervienen ribozimas integradas en la subunidad ribosómica mayor, con la participación directa del ARNr 28 S (eucariotas) o del ARNr 23 S (procariotas).
Recordemos que las ribozimas son filamentos de ARN (en este caso, ARN ribosómico) con capacidad catalítica.
20.- ¿En qué fase de la traducción tiene lugar la formación de los enlaces peptídicos? Explique cómo se establece el primero de ellos.
• Los enlaces peptídicos se forman durante la fase de elongación, gracias a la acción catalítica del complejo enzimático peptidil transferasa. Estos enlaces son los que posibilitan el progresivo alargamiento de la cadena peptídica que se sintetiza.
• Recordemos que el enlace peptídico se establece entre los grupos carboxilo y amino de sendos aminoácidos. Esquema:
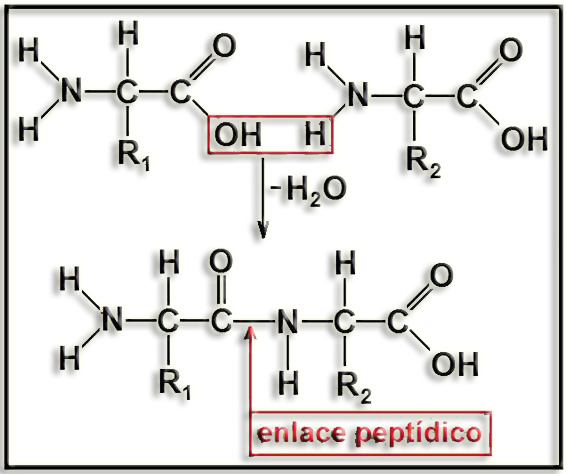
La situación previa para la formación del primer enlace peptídico es que cada aminoácido está unido mediante enlace tipo éster a su ARN transferente, constituyendo el correspondiente aminoacil-ARNt. Esquema:
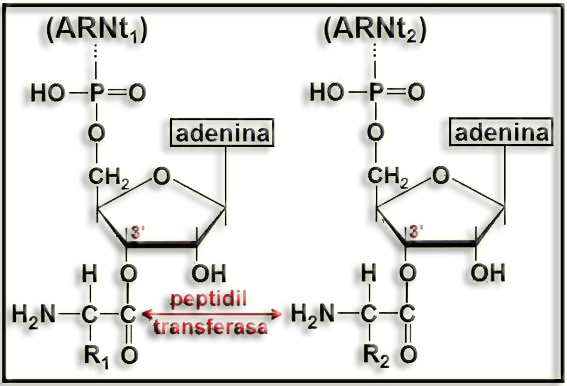
El mecanismo de la reacción está fundamentado en la interacción del grupo amino del 2º aminoácido con el carboxilo del 1º, lo cual implica la rotura del enlace éster que lo une al ARNt1. Esquema:
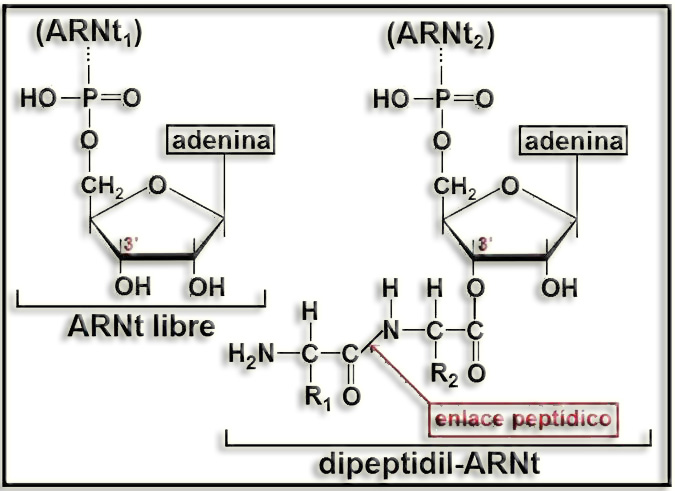
La reacción formadora del enlace peptídico es idéntica durante el transcurso de la biosíntesis proteica. Cabe observar que el primer aminoácido de la cadena peptídica presenta el grupo amino libre (-NH2).
21.- Represente sin fórmulas la formación de los enlaces peptídicos primero y último durante la síntesis proteica.
Sin emplear fórmulas y utilizando la letra A para los aminoácidos, la formación de los enlaces peptídicos durante la biosíntesis de proteínas se puede representar mediante las siguientes expresiones:
• Formación del primero.
(A1)-ARNt(1) + (A2)-ARNt(2) —› ARNt(1) + (A1)-(A2)-ARNt(2)
• Formación del último.
(A1)-(A2)-…-(An-1)-ARNt(n-1) + (An)-ARNt(n) —› ARNt(n-1) + (A1)-(A2)-…-(An-1)-(An)-ARNt(n)
Nota.- También suelen utilizarse subíndices (en lugar de superíndices) para diferenciar a los ARNt: ARNt(1), ARNt (n-1), etc.
22.- ¿Qué representa la estructura molecular adjunta? Identifique las partes numeradas.
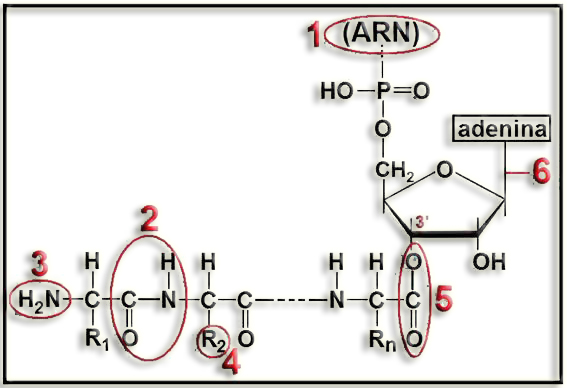
• Esta estructura representa una cadena peptídica formada por n aminoácidos, el último de los cuales está unido al ARN transferente, conjunto que constituye el llamado peptidil-ARNt.
• Partes numeradas:
1 = ARN transferente, específico del último aminoácido
2 = enlace peptídico
3 = grupo amino terminal (propio del 1er aminoácido)
4 = radical o cadena lateral del 2º aminoácido
5 = enlace éster, establecido entre el grupo carboxilo del último aminoácido y el extremo 3’-OH del ARNt.
6 = enlace N-glucosídico (entre la ribosa y la base nitrogenada)
23.- ¿Cómo se separa la cadena peptídica sintetizada del último ARN transferente al que permanece unida? Haga esquemas.
La separación de la cadena peptídica también es llevada a cabo por el complejo enzimático peptidil transferasa, que en este caso final, cuando un factor de liberación ocupa el sitio A del ribosoma (en lugar de otro aminoacil-ARNt), hidroliza el enlace éster que une la cadena peptídica al extremo 3' del ARNt. En otras palabras: la cadena peptídica queda libre al ser "transferida" a una molécula de agua.
Esquema de la situación previa:
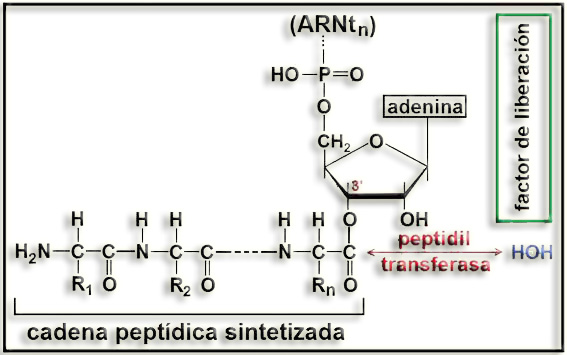
Esquema resultante de la actividad peptidil transferasa:
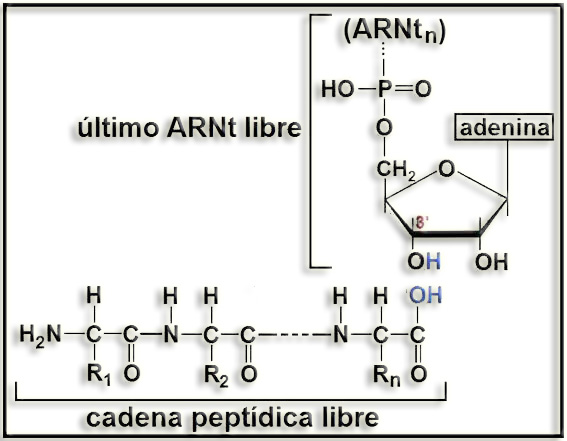
Se observa que el primer aminoácido de la cadena peptídica sintetizada tiene libre el grupo amino, y el último, el carboxilo.
24.- ¿Qué entiende por maduración o procesado de la proteína sintetizada?
Esto significa que la cadena peptídica sintetizada y liberada se pliega adoptando la conformación tridimensional característica para ser funcional (proteína nativa).
Antes o después del plegamiento, dicha cadena puede sufrir algunas transformaciones por acción enzimática, tales como: eliminación del primer aminoácido, incorporación de grupos metilo, carboxilo u otros en los radicales de ciertos aminoácidos, o bien, establecer uniones con oligosacáridos o grupos prostéticos.
Tal serie de transformaciones recibe también la denominación de procesado postraduccional.
25.- (Consulte el código genético). Prediga el resultado y escriba una conclusión acerca del experimento realizado utilizando como molde, en un sistema de síntesis proteica libre de células, el polinucleótido:

Según la clave genética, AAA codifica a la lisina (Lys) y AAC, a la asparagina (Asn). El polipéptido resultante es:
H2N-Lys-(Lys)n-Asn-COOH
Puesto que la asparagina es el residuo carboxilo terminal, el codón AAC debió ser el último en traducirse.
Conclusión: el ARN mensajero se traduce en el sentido 5’ –› 3’.
Nota.- En las células procariotas el ARNm puede ser traducido mientras se completa la transcripción, dado que su extremo 5’ interacciona con los ribosomas instantes después de ser sintetizado.
26.- Si un ARN mensajero está formado por 600 nucleótidos, ¿puede ser posible que la cadena peptídica sintetizada tenga tan solo 60 aminoácidos? Justifique la respuesta.
Considerando que los 600 nucleótidos forman la secuencia codificante del ARNm, cabe esperar que la proteína sintetizada posea 200 aminoácidos, dado que cada uno está codificado por un codón o triplete de nucleótidos.
Si la proteína resultante tiene 60 aminoácidos caben dos posibilidades: que la presencia de un codón mudo o sin sentido interrumpa la síntesis, o bien, que la secuencia codificante conste de 180 nucleótidos, correspondiendo el resto a las regiones que no se traducen (5’-UTR y 3’-UTR).
27.- En relación con la traducción o biosíntesis de proteínas, ordene las siguientes frases de la manera correcta:
• El ribosoma se desplaza un triplete en el sentido 5’ –› 3’.
• Posteriormente se une la subunidad mayor.
• El ARNm se une a la subunidad menor del ribosoma.
• Al lugar A llega otro ARNt con el siguiente aminoácido.
• Ahora el dipéptido se halla en el lugar P (peptidil) y queda libre el lugar A (aminoacil).
• Se unen ambos aminoácidos mediante un enlace peptídico.
La ordenación correcta de las frases anteriores es la siguiente:
• El ARNm se une a la subunidad menor del ribosoma.
• Posteriormente se une la subunidad mayor.
• Al lugar A llega otro ARNt con el siguiente aminoácido.
• Se unen ambos aminoácidos mediante un enlace peptídico.
• El ribosoma se desplaza un triplete en el sentido 5’ –› 3’.
• Ahora el dipéptido se halla en el lugar P (peptidil) y queda libre el lugar A (aminoacil).
28.- ¿Cuál es el destino de las proteínas sintetizadas? ¿A qué se llama péptido señal (secuencia señalizadora)?
• El destino de las proteínas sintetizadas varía según la ubicación de los ribosomas que han intervenido en el proceso:
Ribosomas libres. El destino de las proteínas sintetizadas por los ribosomas libres es alguno de los siguientes: citosol, membrana plasmática, citoesqueleto, mitocondrias, plastos, peroxisomas, núcleo (histonas, polimerasas, etc.).
Ribosomas del RER. El destino de las proteínas sintetizadas por ribosomas asociados al retículo endoplasmático rugoso es: el complejo de Golgi y los lisosomas.
• Por lo que respecta al péptido señal (o secuencia señalizadora), las proteínas recién sintetizadas suelen presentar uno o varios de ellos, que sirven para determinar su ubicación posterior en función de los receptores específicos de la membrana de los orgánulos. La ausencia de péptido señal determina que la proteína quede en el citosol.
29.- Escriba un comentario sobre el esquema adjunto, indicando el significado de las figuras numeradas y de las letras A y B.
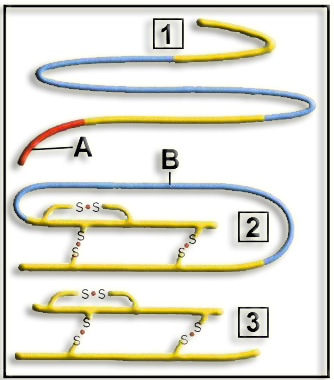
Este esquema representa la maduración de la insulina.
• La figura 1 corresponde a la preproinsulina, observándose en su cadena peptídica 4 regiones diferenciadas, siendo “A” la secuencia señal (color rojo).
• La figura 2 es la proinsulina, formada cuando se pierde la secuencia señal del extremo N-terminal. La letra B está marcando la parte denominada péptido C (color azul), que será eliminado por un doble corte proteolítico.
• La figura 3 representa el estado final, es decir, la insulina madura, plegada y funcional, observándose los tres puentes disulfuro característicos, dos intercatenarios y uno intracatenario.
Nota.- La insulina es la hormona hipoglucémica que se produce en el páncreas, concretamente, en las células beta de los islotes de Langerhans, en forma de un precursor inactivo.
La insulina activa está integrada por 51 aminoácidos, 30 en la cadena B y 21 en la A, siendo ésta reconocible por su puente disulfuro intracatenario. La secuencia fue descubierta por el bioquímico inglés F. Sanger, que fue galardonado con un premio Nobel.
La deficiencia de insulina desencadena la diabetes mellitus.
30.- ¿Por qué se dice que los ARN mensajeros presentan inestabilidad metabólica?
Se dice que los ARN mensajeros presentan inestabilidad metabólica porque su existencia en el interior de la célula es limitada, al ser degradados por nucleasas específicas. Tal circunstancia implica que debe existir una síntesis frecuente de los ARNm, lo cual requiere una transcripción activa del gen correspondiente.
En las células procariotas, el transcrito primario de un gen sirve como ARNm y suele tener una vida media muy corta (minutos). Sin embargo, el periodo funcional de los ARNm eucariotas es mayor, puesto que la caperuza y la cola adquiridas durante el procesado postranscripcional parecen conferir cierta protección frente a las nucleasas.




