Traducción 3 (soluciones)
1.- Interprete el esquema adjunto y nombre las partes numeradas.
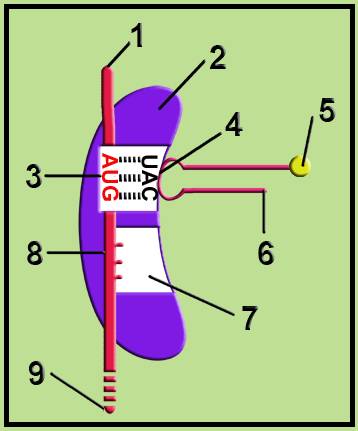
• Este esquema sirve para ilustrar la fase de iniciación del proceso de la traducción. El llamado complejo de iniciación está aún incompleto ya que no ha sido dibujada la subunidad ribosómica mayor.
• Los nombres de las partes numeradas son:
1 = extremo 5’ del ARN mensajero
2 = subunidad ribosómica menor
3 = codón iniciador (5’-AUG-3’)
4 = anticodón (3’-UAC-5’) del ARNt iniciador.
5 = aminoácido unido al extremo aceptor (3’) del ARNt, conjunto que constituye un aminoacil-ARNt. Dada la presencia del codón AUG, podría tratarse de la metionina (eucariotas) o la formilmetionina (procariotas).
6 = extremo 5’ del ARNt iniciador
7 = sitio o lugar A (aminoacilo)
8 = segundo codón del ARNm
9 = extremo 3’ del ARNm
2.- Interprete el esquema adjunto indicando el paso inmediato posterior. Nombre las partes numeradas. ¿Cuántos enlaces como el señalado con el nº 7 se formarían?
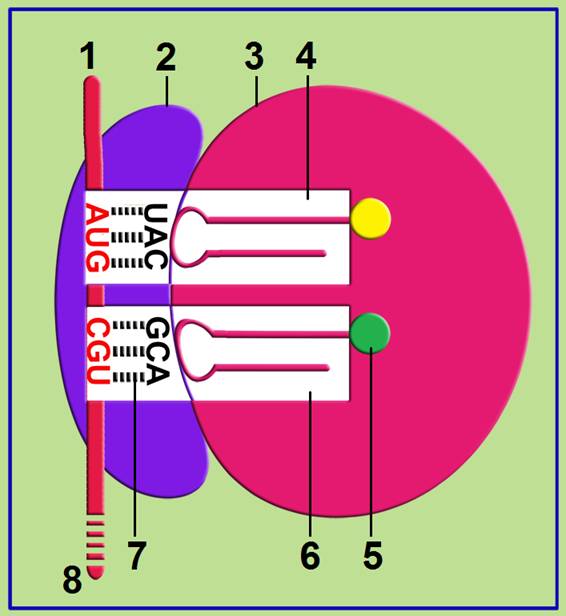
• Este esquema representa el comienzo de la elongación. Se observa que el complejo de iniciación está formado y que el siguiente aminoacil-ARNt (con el anticodón GCA) ha ocupado el sitio A.
• El paso inmediato posterior es la formación del primer enlace peptídico, que se establece entre el primer aminoácido (color amarillo) y el segundo (verde), según reacción catalizada por el complejo enzimático peptidil transferasa.
• Los nombres de las partes numeradas son:
1 = extremo 5’ del ARN mensajero
2 = subunidad ribosómica menor
3 = subunidad ribosómica mayor
4 = sitio P, ocupado por el ARNt iniciador
5 = arginina (según el codón 5’-CGU-3’). Cada aminoácido se halla unido al extremo aceptor de su ARNt específico.
6 = sitio A, ocupado por el arginil-ARNtArg.
7 = enlace por puente de hidrógeno (pph)
8 = extremo 3’ del ARN mensajero
• La interacción codón-anticodón se produce por apareamiento antiparalelo, mediante la formación de enlaces por puente de hidrógeno (pph), entre las cadenas de ARNm y de ARNt.
Dado que se forman 2 enlaces pph entre el par AU, y 3 entre el GC, se formarían 7 enlaces entre 5’-AUG-3’ y 3’-UAC-5’. En el segundo caso, entre 5’-CGU-3’ y 3’-GCA-5’, se formarían 8.
3.- Interprete el esquema adjunto y nombre las partes numeradas.
• Este esquema representa la fase de iniciación. La subunidad ribosómica menor se ha desplazado por el ARN mensajero (en sentido 5’ --> 3’), con el aporte energético del ATP, hasta encontrar el codón iniciador, el cual queda emparejado con el anticodón del ARNt iniciador (que en este dibujo está situado en la zona del sitio P).
• Partes numeradas:
1 = caperuza 5’ del ARNm
2 = extremo 3’ del ARNm
3 = región que no se traduce (5’-UTR)
4 = enlace tipo éster
5 = aminoacil-ARNt iniciador
6 = zona parcial del sitio P
7 = zona parcial del sitio A
4.- Interprete el esquema adjunto e identifique las partes numeradas.
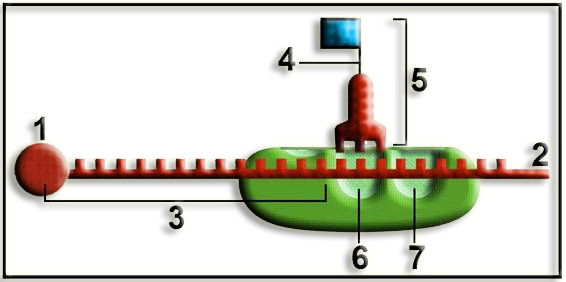
• Este esquema ilustra el comienzo de la elongación, justo antes de la formación del primer enlace peptídico.
• Partes numeradas:
1 = caperuza 5’ del ARNm (propia de los ARNm eucarióticos)
2 = extremo 3’ del ARNm
3 = subunidad ribosómica menor (40 S)
4 = sitio E (exit)
5 = subunidad ribosómica mayor (60 S)
6 = primer aminoácido (metionina en la biosíntesis eucariótica)
7 = siguiente aminoácido
8 = enlace covalente, tipo éster
9 = sitio P, ocupado por el aminoacil-ARNt iniciador: ARNtMet
10 = sitio A, ocupado por el siguiente aminoacil-ARNt
5.- Haga una interpretación del esquema adjunto y nombre las partes numeradas.
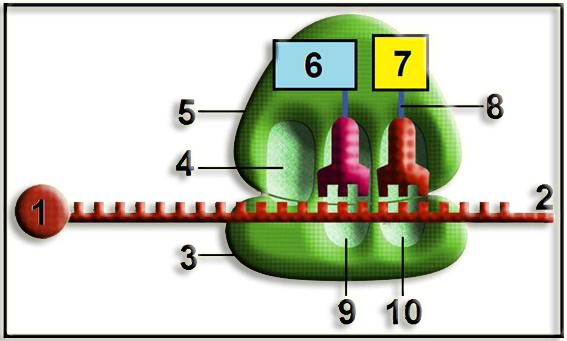
• Este esquema ilustra la fase de elongación, tras la formación del primer enlace peptídico. También se observa que el ribosoma, concretamente la subunidad mayor, ha realizado la transposición (o translocación).
• Partes numeradas:
1 = región que no se traduce (5’-UTR)
2 = caperuza 5’ del ARNm
3 = ARNt libre o saliente, situado en el sitio E (exit)
4 = subunidad ribosómica mayor (translocada)
5 = primer aminoácido (metionina)
6 = segundo aminoácido
7 = enlace peptídico
8 = enlace éster
9 = zona del sitio A en la subunidad mayor
10 = dipeptidil-ARNt
11 = extremo 3’ del ARNm
6.- Interprete el esquema adjunto e indique el significado de los números y de las letras.
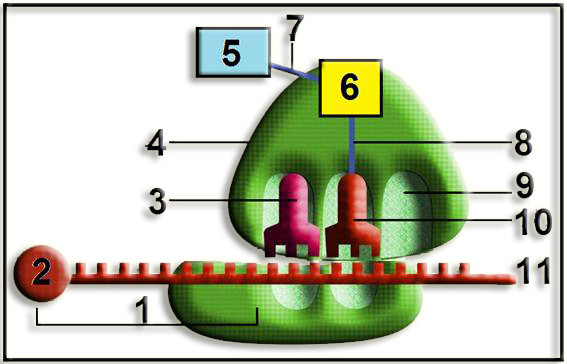
• Este esquema ilustra la fase de elongación. Se observa que la cadena peptídica consta de 4 aminoácidos y está a punto de ser incorporado el 5º.
• Los números representan aminoácidos, de los cuales los 4 primeros ya están unidos.
• Partes marcadas con letras:
A = ARNt saliente (estaba unido al aminoácido nº 3)
B = sitio E (exit)
C = enlace peptídico
D = enlace éster
E = peptidil-ARNt (en el sitio P)
F = aminoacil-ARNt (en el sitio A)
G = subunidad ribosómica menor
H = extremo 3’ del ARN mensajero
7.- Interprete el esquema adjunto e indique el significado de los números y de las letras.
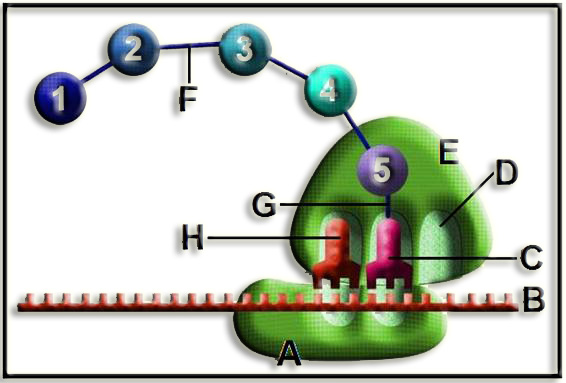
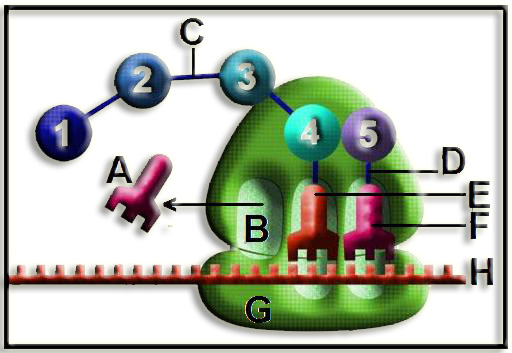
• Este esquema ilustra la fase de elongación. Se observa que la cadena peptídica consta de 5 aminoácidos y que ha tenido lugar un desplazamiento de la subunidad ribosómica mayor.
• Los números representan aminoácidos, de los cuales los 5 primeros ya están unidos.
• Partes marcadas con letras:
A = subunidad ribosómica menor
B = extremo 3’ del ARN mensajero
C = peptidil-ARNt (en el sitio P)
D = sitio A (que será ocupado por el siguiente aminoacil-ARNt cuando avance la subunidad menor para completar la translocación)
E = subunidad ribosómica mayor
F = enlace peptídico
G = enlace éster
H = ARNt saliente (que estaba unido al aminoácido nº 4), situado en el sitio E.
8.- Interprete el esquema adjunto e identifique las partes numeradas.
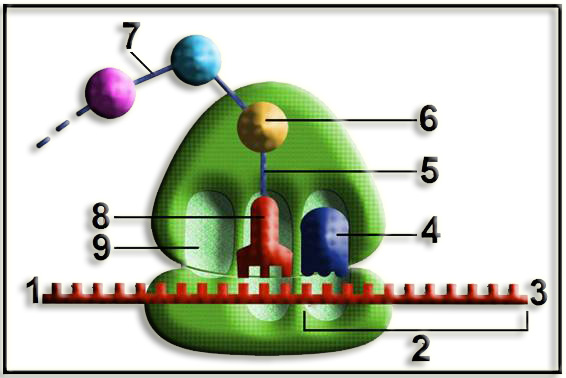
• Este esquema representa el comienzo de la fase de terminación. Cuando aparece en el ARNm un codón “stop” o sin sentido, que señala el fin de la traducción, el sitio A del ribosoma es ocupado por un factor proteico de terminación.
• Partes numeradas:
1 = ARN mensajero
2 = región que no se traduce (3’-UTR)
3 = extremo 3’ del ARNm
4 = factor de liberación o terminación, ocupando el sitio A
5 = enlace éster
6 = último aminoácido de la cadena sintetizada
7 = enlace peptídico
8 = peptidil-ARNt (alojado en el sitio P)
9 = sitio E
9.- ¿Qué representa el esquema adjunto? Identifique las partes numeradas.
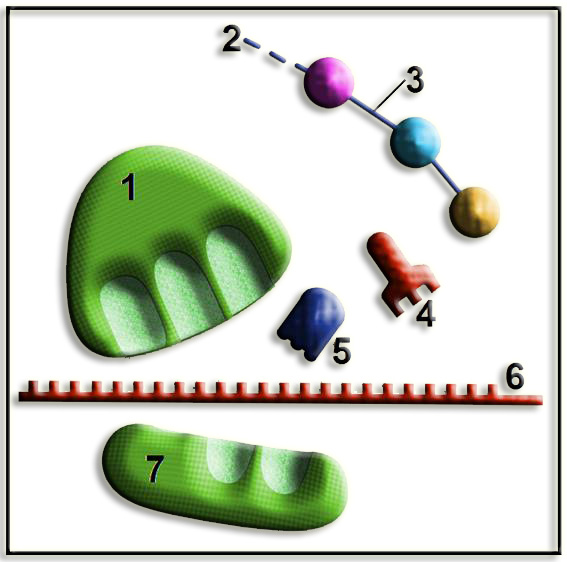
• Este esquema representa la terminación de la traducción, que se caracteriza por la separación de los componentes que han intervenido en el proceso.
• Partes numeradas:
1 = subunidad ribosómica mayor
2 = cadena peptídica sintetizada
3 = enlace peptídico
4 = ARN transferente
5 = factor de liberación
6 = ARN mensajero
7 = subunidad ribosómica menor
10.- Cite la fase que representa el esquema adjunto. Nombre las partes numeradas. (Consulte el código genético).
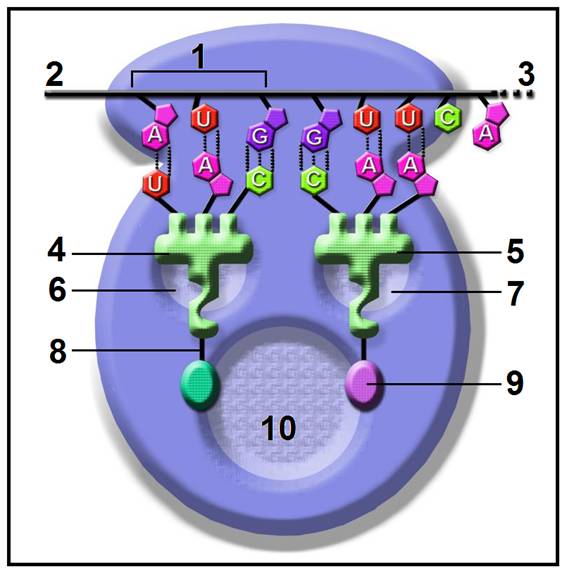
• Este esquema representa el comienzo de la elongación.
• Partes numeradas:
1 = codón iniciador (5’-AUG-3’)
2 = extremo 5’ del ARNm
3 = extremo 3’ del ARNm
4 = ARNt iniciador portando el aminoácido metionina (eucariotas) o formilmetionina (procariotas).
5 = aminoacil-ARNt, concretamente, valil-ARNtV
6 = sitio P
7 = sitio A
8 = enlace tipo éster
9 = valina (según el codón 5’-GUU-3’)
10 = sitio o centro peptidil transferasa
11.- ¿Qué es el “centro peptidil transferasa”?
El sitio o centro peptidil transferasa es una cavidad de la subunidad ribosómica grande, en la cual se desarrolla la actividad catalítica de polimerización de aminoácidos, es decir, la formación de cada uno de los enlaces peptídicos, acción llevada a cabo por el llamado complejo enzimático peptidil transferasa.
Este complejo enzimático posee varios componentes proteicos, pero en la catálisis intervienen ribozimas integradas en el ARN ribosómico de la subunidad mayor, concretamente, el ARNr 28 S (eucariotas) o el ARNr 23 S (procariotas).
12.- ¿Cuáles son los diversos “sitios” que suelen considerarse en el ribosoma?
Se consideran los siguientes:
• Sitio A (aminoacilo o aceptor), donde entra el aminoacil-ARNt en cada ciclo de elongación.
• Sitio P (peptidilo o donador), en el cual se aloja el peptidil-ARNt.
• Sitio E (de eyección o salida), que está ocupado por el ARNt que ha quedado libre.
• Sitio peptidil transferasa, que se halla en la subunidad mayor, cerca del enlace que une los ARNt con su péptido y su aminoácido. En este sitio está localizado el complejo enzimático peptidil transferasa (cuya actividad catalítica reside en el ARN ribosómico).
13.- Interprete el esquema adjunto y nombre las partes numeradas.
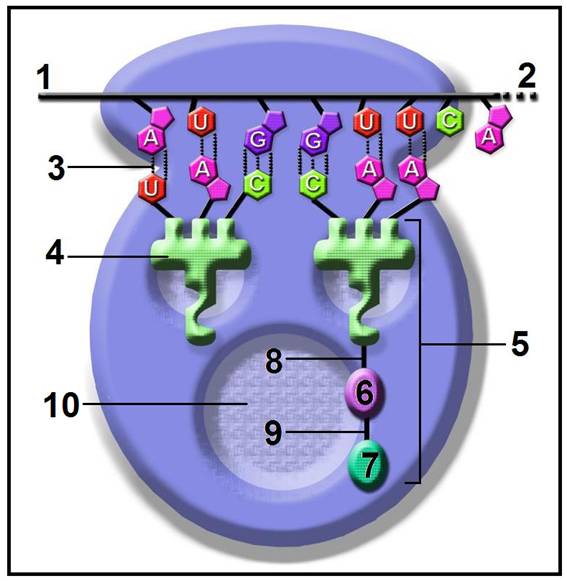
• Este esquema ilustra la fase de elongación, justo después de formarse un enlace peptídico entre el aminoácido que estaba unido al ARNt del sitio P y el del sitio A.
• Partes numeradas:
1 = extremo 5’ del ARNm
2 = extremo 3’ del ARNm
3 = enlace por puente de hidrógeno
4 = ARNt iniciador sin el aminoácido que portaba
5 = dipeptidil-ARNt Val
6 = valina (según el codón 5’-GUU-3’)
7 = metionina (eucariotas) o formilmetionina (procariotas)
8 = enlace tipo éster
9 = enlace peptídico
10 = sitio o centro peptidil transferasa
14.- Interprete el esquema adjunto e identifique las partes numeradas.
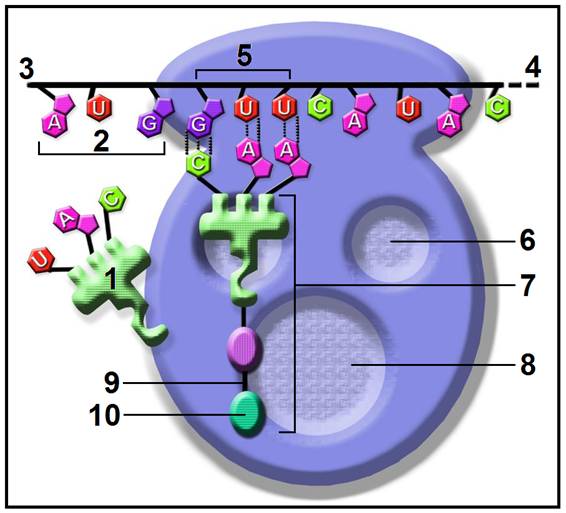
• Este esquema ilustra la fase de elongación, observándose que ha tenido lugar la transposición (translocación), que se caracteriza por el avance del ribosoma tres nucleótidos en el ARNm, en el sentido 5’ —> 3’.
• Partes numeradas:
1 = ARNt iniciador libre o saliente (en este esquema no está representado el sitio E).
2 = codón iniciador
3 = extremo 5’ del ARNm
4 = extremo 3’ del ARNm
5 = segundo codón: 5’-GUU-3’
6 = sitio A libre (que instantes después será ocupado por el siguiente aminoacil-ARNt)
7 = dipeptidil-ARNt Val (Met-Val-ARNtV), alojado en el sitio P (antes de la translocación estaba en el sitio A)
8 = sitio del complejo enzimático peptidil transferasa
9 = enlace peptídico
10 = metionina (eucariotas) o formilmetionina (procariotas)
15.- Interprete el esquema adjunto y nombre las partes numeradas. (Consulte la clave genética).
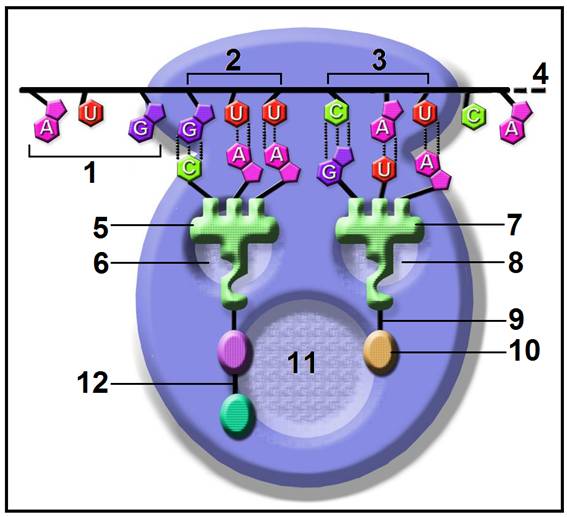
• El esquema representa la fase de elongación. Se observa que el dipeptidil-ARNt está alojado en el sitio P y que el lugar A está ocupado por otro aminoacil-ARNt.
• Partes numeradas:
1 = codón iniciador: 5’-AUG-3’
2 = segundo codón: 5’-GUU-3’
3 = tercer codón: 5’-CAU-3
4 = extremo 3’ del ARNm
5 = dipeptidil-ARNt Val
6 = sitio P
7 = histidil-ARNtH
8 = sitio A
9 = enlace tipo éster
10 = histidina (según el codón 5’-CAU-3’)
11 = sitio o centro peptidil transferasa
12 = enlace peptídico
16.- Interprete el esquema adjunto y nombre las partes numeradas. (Consulte la tabla del código genético).
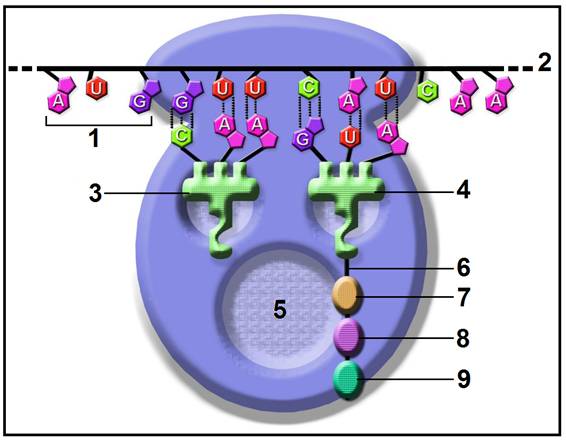
• Este esquema representa la fase de elongación. Se observa que el tripeptidil-ARNt está alojado en el sitio A y que el lugar P está ocupado por el ARNt cuyo aminoácido ha sido adicionado a la cadena peptídica que se está sintetizando. El tripeptidil-ARNt, cuando tenga lugar otra translocación, volverá a ocupar el sitio P.
• Partes numeradas:
1 = codón iniciador (5’-AUG-3’)
2 = extremo 3’ del ARNm
3 = ARNt específico de la valina (ARNtV)
4 = tripeptidil-ARNtH, concretamente: Met-Val-His-ARNtH.
5 = sitio o centro peptidil transferasa
6 = enlace tipo éster
7 = histidina (según el codón 5’-CAU-3’)
8 = valina (según el codón 5’-GUU-3’)
9 = metionina (según el codón 5’-AUG-3’)
17.- Escriba un comentario sobre el esquema adjunto e identifique las partes numeradas.
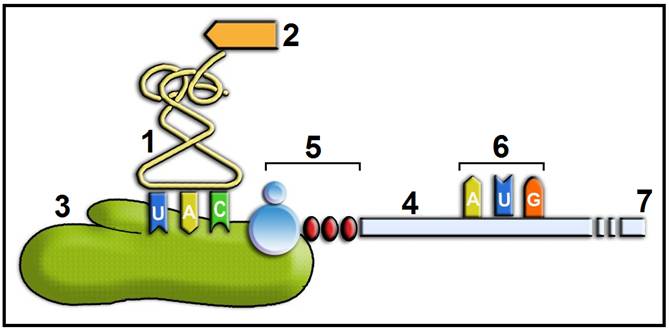
• Este esquema representa la primera fase de la traducción, concretamente, el comienzo de la iniciación. Se observa la unión de la subunidad ribosómica menor (40 S) con el ARN transferente iniciador, portador del primer aminoácido. Además, dicha subunidad interacciona con la caperuza del ARN mensajero.
• Partes numeradas:
1 = ARNt iniciador (con el anticodón 3’-UAC-5’)
2 = metionina (Met, M)
3 = subunidad ribosómica menor (40 S)
4 = ARN mensajero (ARNm)
5 = caperuza de metil guanosina trifosfato, propia de los ARN mensajeros eucarióticos (extremo 5’)
6 = codón iniciador (5’-AUG-3’)
7 = extremo 3’ del ARNm
Nota.- El proceso representado requiere factores de iniciación y aporte energético (GTP).
18.- Interprete el esquema adjunto e identifique las partes numeradas. ¿Qué aminoácidos estarían en las posiciones tercera y última? (Consulte el código genético).
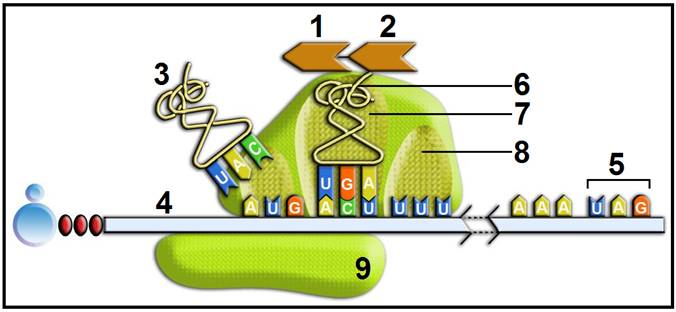
• Este esquema corresponde a la fase de elongación. Se observa que ya se ha formado el enlace peptídico entre los dos primeros aminoácidos y que está teniendo lugar el desplazamiento del ribosoma (transposición, translocación), el cual avanza exactamente tres nucleótidos en el sentido 5’ --> 3’, si bien la subunidad mayor se mueve poco antes que la menor, proceso que requiere aporte energético (GTP) y un factor de elongación.
• Partes numeradas:
1 = metionina (codificada por el triplete AUG)
2 = treonina (codificada por el triplete ACU)
3 = liberación del ARNtMet desde el sitio E (exit)
4 = ARN mensajero eucariótico (con caperuza 5’)
5 = codón mudo o sin sentido (“stop” de la traducción)
6 = dipeptidil-ARNt (concretamente: Met-Thr-ARNtThr)
7 = sitio P, ocupado por el dipeptidil-ARNt
8 = sitio A, que será ocupado por el siguiente aminoacil-ARNt (no representado).
9 = subunidad ribosómica menor (40 S)
• El tercer aminoácido sería la fenilalanina, según el codón UUU. Esto pasaría cuando quede completada la transposición por el avance de la subunidad menor, de forma que el sitio A sea ocupado por el siguiente aminoacil- ARNt (fenilalanil-ARNtF).
• El último aminoácido de la cadena sintetizada sería la lisina, dado que el último codón (que antecede a la señal de “stop”) es AAA.
19.- ¿Qué representa el esquema adjunto? Nombre las partes numeradas. ¿Qué significado tienen las letras UAC?
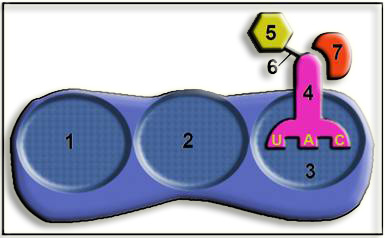
• Este esquema representa el comienzo de la fase de iniciación, observándose que la subunidad ribosómica menor se ha asociado, en el sitio o lugar A, con el aminoacil-ARNt iniciador. Esta asociación requiere la intervención de un factor de iniciación y el aporte energético del GTP.
• Partes numeradas:
1 = sitio o lugar E
2 = sitio o lugar P
3 = sitio o lugar A
4 = ARNt iniciador “cargado” (aminoacil-ARNti)
5 = aminoácido iniciador: metionina (eucariotas) o formilmetionina (procariotas)
6 = enlace tipo éster
7 = factor de iniciación
• Las letras UAC representan el anticodón del ARNt iniciador, que posteriormente se emparejará con el codón AUG. Estos emparejamientos tienen carácter antiparalelo, por lo que la polaridad de los tripletes citados será: 3’-UAC-5’ y 5’-AUG-3’.
20.- Interprete el esquema adjunto indicando cómo se ha formado el conjunto representado. Nombre las partes numeradas.
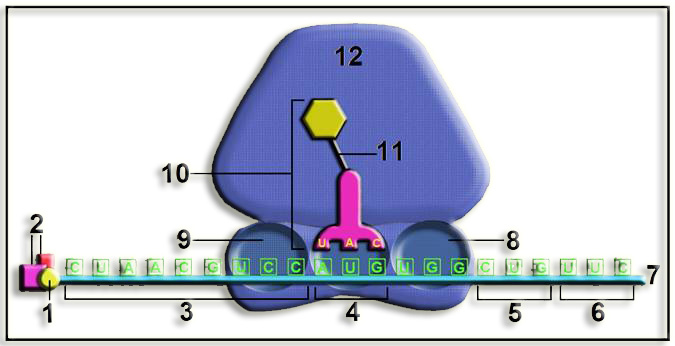
• Este esquema representa la formación completa del complejo de iniciación en eucariotas (por la presencia de caperuza en el ARNm).
• La subunidad ribosómica menor, unida con el metionil-ARNt, interacciona con factores de iniciación y la caperuza del ARNm, desplazándose por esta cadena, con el aporte energético del ATP, hasta que el anticodón UAC reconoce al codón iniciador, AUG. Entonces se une la subunidad mayor constituyendo el conjunto denominado complejo de iniciación 80 S, que se mueve tres nucleótidos en el sentido 5’ --> 3’, de forma que el metionil-ARNt iniciador (que estaba en el sitio A) queda alojado en el sitio P. Se observa que en este preciso momento los sitios E y A están libres.
• Partes numeradas:
1 = caperuza 5’ del ARNm
2 = factores de iniciación
3 = región que no se traduce (5’-UTR o líder)
4 = codón iniciador (5’-AUG-3’), situado en el lugar P. Se observa que dicho triplete queda emparejado con el anticodón 3’-UAC-5’.
5 = codón 3º (5’-CUG-3’)
6 = codón 4º (5’-UUC-3’)
7 = extremo 3’ del ARNm
8 = sitio o lugar A
9 = sitio o lugar E
10 = aminoacil-ARNt, concretamente: metionil-ARNtM
11 = enlace tipo éster
12 = subunidad ribosómica mayor (60 S)
21.- Escriba un comentario sobre el esquema adjunto y nombre las partes numeradas. (Consulte el código genético).
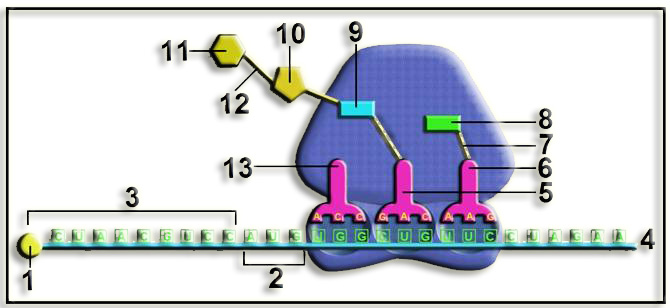
• Este esquema representa la fase de elongación, observándose que los sitios E, P y A están ocupados por sendos ARNt. Los sucesivos aminoácidos que se van añadiendo a la cadena quedan unidos mediante un enlace peptídico (gracias a la acción del complejo enzimático peptidil transferasa). En este momento la cadena consta de 3 aminoácidos y está a punto de ser adicionado el 4º.
• Partes numeradas:
1 = caperuza 5’ del ARNm
2 = codón iniciador (5’-AUG-3’)
3 = región que no se traduce (5’-UTR o líder)
4 = extremo 3’ del ARNm
5 = tripeptidil-ARNt, concretamente: Met-Trp-Leu-ARNtLeu. Este ARNt ocupa el sitio P.
6 = aminoacil-ARNt, concretamente: fenilalanil-ARNtF. Ocupa el sitio A.
7 = enlace tipo éster
8 = fenilalanina (según el codón UUC)
9 = leucina (según el codón CUG)
10 = triptófano (según el codón UGG)
11 = metionina (según el codón AUG)
12 = enlace peptídico
13 = ARNtTrp libre o saliente, alojado en el sitio E (exit)
22.- ¿En qué consiste la transposición (o translocación)?
Consiste en el desplazamiento del ribosoma, que avanza exactamente 3 nucleótidos por el ARNm, en el sentido 5’à 3’. Las subunidades ribosómicas no se trasladan a la vez, sino que en primer lugar avanza la subunidad mayor y, seguidamente, la menor. Para ello se necesita un factor de elongación y el aporte energético del GTP.
La translocación provoca que el ARNt descargado del sitio P vaya al sitio E, y el ARNt portador del péptido en el sitio A, se mueva al P.
23.- ¿Es cierto que en determinados momentos el peptidil-ARNt ocupa el sitio A en lugar del sitio P? Justifique la respuesta.
Eso es cierto, pero justo antes de la translocación, pues el péptido del peptidil-ARNt (alojado en el sitio P) se transfiere al aminoacil-ARNt, que ocupa el sitio A. Cuando se completa el ciclo de elongación y tiene lugar la translocación, el peptidil-ARNt queda situado en el sitio P, dejando libre el sitio A para la entrada del siguiente aminoacil-ARNt.
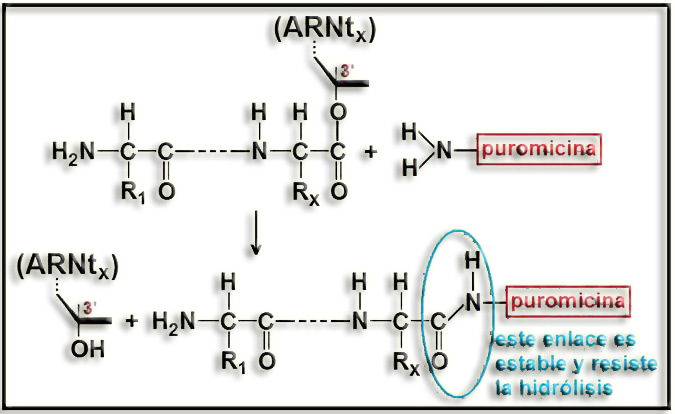
24.- En un libro de 2º de Bachillerato aparece la ilustración adjunta. Escriba un comentario e indique qué representa el compuesto numerado (“1”). Haga un esquema del enlace que se ha formado.
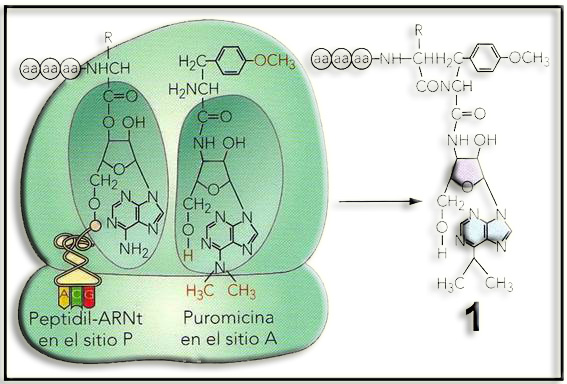
• La biosíntesis de proteínas es inhibida por muchos antibióticos, uno de los cuales es la puromicina, que tiene una estructura similar a un aminoacil-ARNt, lo que le permite ocupar el sitio A de los ribosomas (procariotas y eucariotas).
Seguidamente, el complejo enzimático peptidil transferasa cataliza la transferencia de la cadena peptídica, no a otro aminoácido, sino a la puromicina, formando “peptidil-puromicina”, lo cual provoca la terminación prematura de la síntesis proteica.
• El compuesto marcado con el nº 1 se refiere al complejo peptidil-puromicina, que no al no poder adicionar nuevos aminoácidos determina la interrupción de la síntesis.
• Se ha formado un enlace peptídico al interaccionar el grupo amino de la puromicina con el carboxilo del último aminoácido del peptidil-ARNt sintetizado hasta ese momento.
Esquema:
25.- Interprete el esquema adjunto y nombre las partes numeradas (consulte la clave genética). ¿Qué significado tiene el triplete AAG?
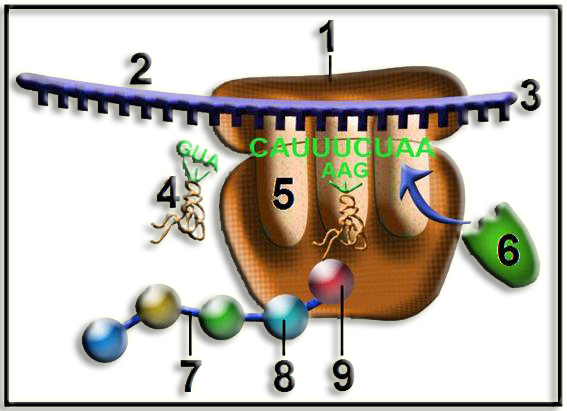
• Este esquema representa el final de la elongación y el comienzo de la terminación. El ribosoma se desplaza por el ARNm, en sentido 5’à3’, hasta que encuentra un codón “stop” o sin sentido (en este caso, UAA). Entonces se incorpora en el sitio A un factor de terminación provocando que se detenga la síntesis de la cadena peptídica.
• Partes numeradas:
1 = subunidad ribosómica menor
2 = ARN mensajero
3 = extremo 3’ del ARNm
4 = ARNt saliente
5 = sitio E (exit)
6 = factor de liberación o terminación
7 = enlace peptídico
8 = histidina (penúltimo aminoácido, codificado por CAU)
9 = fenilalanina (último aminoácido, codificado por UUC)
• El triplete AAG es el anticodón del ARNtF, situado frente al codón UUC.
Recordemos que la interacción codón-anticodón tiene lugar por apareamiento antiparalelo, mediante la formación de enlaces por puente de hidrógeno (en este caso se formarían 7: dos entre cada par AU y tres entre GC).
En el sitio P se observa que el ARNtF es portador de la cadena sintetizada, conjunto que constituye el peptidil-ARNtF.
26.- Interprete el esquema adjunto y nombre las partes numeradas.
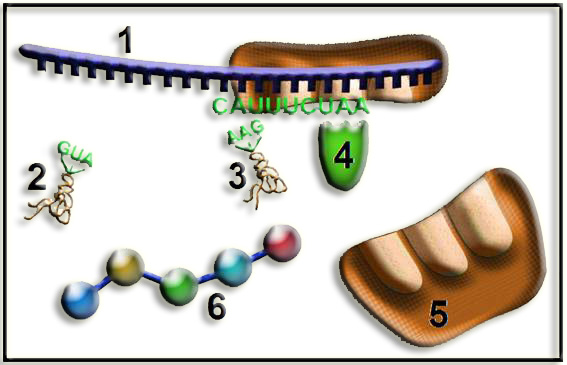
• Este esquema representa la terminación del proceso de la traducción, fase mediada por factores de liberación (ver nota), dando como resultado la separación de los componentes que han intervenido en dicho proceso. Las subunidades ribosómicas pueden reiniciar otro ciclo de traducción.
• Partes numeradas:
1 = ARN mensajero
2 = ARNt libre
3 = ARNt libre
4 = factor de liberación
5 = subunidad ribosómica mayor
6 = cadena peptídica sintetizada (suelen tener numerosos aminoácidos)
Nota.- Los factores de liberación se designan con las siglas RF (del inglés, release factor) y son los que interactúan con los codones de terminación (el RF1, con UAA o UAG, y el RF2, con UAA o UGA).
Asimismo, dichos factores inducen la liberación de la cadena peptídica, la cual abandona el ribosoma por medio de un túnel que atraviesa la subunidad grande y que comienza en la cavidad peptidil transferasa.
27.- ¿Son funcionales las cadenas peptídicas recién sintetizadas?
En general, los polipéptidos recién sintetizados no son funcionales, ya que deben adquirir la conformación tridimensional adecuada para desempeñar su actividad.
Las chaperonas son proteínas que posibilitan el plegamiento correcto de la cadena peptídica recién sintetizada.
La actuación de las chaperonas es importante puesto que, generalmente, las proteínas recién sintetizadas son incapaces por sí solas de alcanzar el plegamiento tridimensional para ser funcionales (conformación nativa).
Las chaperonas se descubrieron a finales de la década de 1970 y, actualmente, las más conocidas son aquellas cuya masa molecular se aproxima a 60 mil unidades de masa atómica, o sea, 60 kDa, que suelen designarse como chaperoninas.
Cabe advertir que las chaperoninas no son enzimas, pues no modifican covalentemente al sustrato con el que interaccionan, sino que actúan como moldes, facilitando que el polipéptido desplegado pueda alcanzar su estructura tridimensional nativa.
28.- ¿Son perdurables las proteínas celulares?
No, ya que la vida media de las proteínas varía desde unos minutos a varios días.
Las ciclinas, que regulan el ciclo celular, son degradadas rápidamente. Por el contrario, las tubulinas que forman los microtúbulos tienen una vida más larga.
La actividad celular requiere que todas las proteínas sean renovadas continuamente. A este respecto cabe señalar que, en las células procariotas, la velocidad de la síntesis proteica oscila en torno a 15 aminoácidos por segundo, siendo menor en las eucariotas, ya que se suelen adicionar de 2 a 5 aminoácidos por segundo.
Las proteínas más o menos defectuosas al término de su período funcional, así como las que muestran un plegamiento incorrecto, deben ser eliminadas para no interferir con el funcionamiento celular. En las células eucariotas, tal eliminación ocurre en los lisosomas y en los proteasomas.
29.- ¿Qué son los proteasomas (o proteosomas)?
Los proteasomas son agregados de múltiples subunidades proteicas, que delimitan una zona cilíndrica central, llamada cámara proteolítica, cuya función es degradar proteínas defectuosas o de vida corta, para lo cual se requiere el aporte energético del ATP.
Las proteínas que deben ser destruidas han sido previamente “ubiquitinadas”. La adición de varias moléculas de ubiquitina, un polipéptido de 76 aminoácidos, constituye la señal reconocible por los complejos moleculares de los proteasomas para introducirlas en la cámara proteolítica.
30.- Explique el significado de esta expresión: “síntesis no ribosómica de péptidos” (ilustre con esquemas).
Cuando la actividad celular requiere sintetizar cadenas peptídicas muy cortas, llamadas oligopéptidos, no se pone en marcha la traducción ribosómica de ningún ARNm, sino que se activa en el citosol un mecanismo diferente y mucho más simple, que posibilita la formación de enlaces peptídicos entre los aminoácidos sucesivamente incorporados.
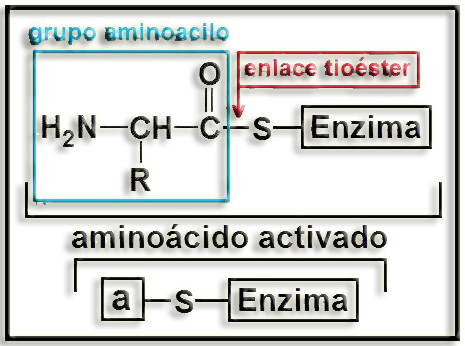
La acción enzimática que regula este proceso requiere ATP y se inicia una vez originados los diversos aminoacil-AMP, siendo posteriormente activados los aminoácidos por la formación de “tioésteres” unidos a la enzima.
Esquema (aminoácido = a):
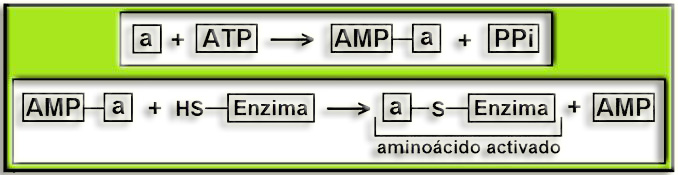
El “tioéster” se forma al unirse el grupo carboxilo con el sulfhidrilo de la enzima, es decir:
…-COOH + HS-E --> …-CO-S-E + HOH.
Esquema:
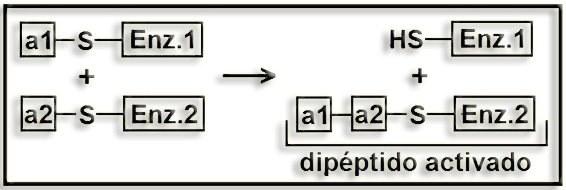
Posteriormente tiene lugar la transferencia del primer aminoácido al siguiente, mediante la formación de un enlace peptídico, resultando un dipéptido “activado”, esto es, capaz de adicionar otro aminoácido.
Esquema:
Si se representa la formación del enlace peptídico resulta este otro esquema:
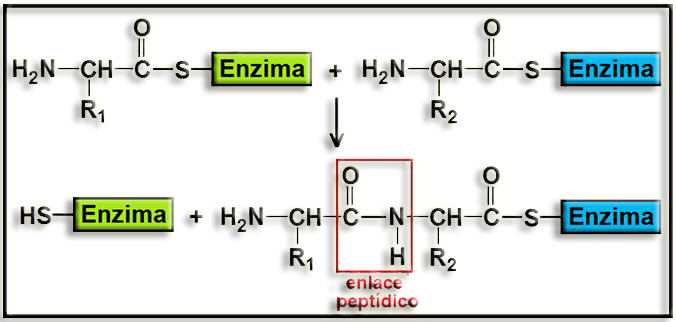
Esta acción enzimática se va repitiendo hasta constituir el oligopéptido.
Las reacciones anteriores se pueden resumir así:
•) aminoácido + ATP —› aminoacil-AMP + PPi
•) aminoacil-AMP + HS-E —› aminoacil-S-E + AMP
Poniendo “(a)-S-E” en lugar de “aminoacil-S-E”, resulta:
•) (a1)-S-E + (a2)-S-E’ —› (a1)-(a2)-S-E’ + HS-E




